Aplicando ingeniería reversa a David Goodsell
Introducción
Desde hace un tiempo que estoy interesado en la relación existente entre el arte y la biología estructural. En especial, me atraen las diversas formas de representar las estructuras tridimensionales de las macromoléculas biológicas, ya sea mediante gráficos computacionales, animaciones o modelos físicos tangibles.
Al igual que mucha gente, me gusta el estilo único con que David Goodsell ilustra las estructuras de la sección Molecule of the Month (MotM) en el Protein Data Bank. Sin embargo, reproducir su aspecto es más complicado de lo que parece. Para generar las figuras de MotM, Goodsell usa software personalizado (aparentemente en Fortran, según este link en alemán). Existen varios tutoriales y herramientas en Internet para emular su apariencia1, pero ninguno de esos procedimientos me convence totalmente. Sin embargo, tomé algunas ideas de ellos y logré elaborar dos métodos usando PyMOL. Al principio solo quería averiguar cómo hacer figuras con el estilo de Goodsell, pero finalmente se transformó en una suerte de obsesión. Espero que les sea útil.
Una manera fácil usando solo PyMOL
Para empezar, voy a reproducir la figura que aparece en la entrada de MoTM del mes de septiembre del año 2000, correspondiente a la lisozima de clara de huevo de gallina (PDB ID: 2LYZ).
Primero hay que cargar la estructura de 2LYZ (por ejemplo usando fetch 2lyz), para luego remover las aguas con remove solvent, mostrar la proteína como esferas usando show spheres, poner un fondo blanco con bg_color white y coloreando por elemento de acuerdo a su esquema clásico (carbonos blancos, nitrógenos, oxígenos y azufres en tonos azulados, rojizos y amarillentos, respectivamente):2
color white
color lightblue, name N*
color blue, resn ARG+LYS and sidechain and name N*
color salmon, name O*
color red, resn GLU+ASP and sidechain and name O*
color sulfur, name S*
Una vez terminada la preparación de la escena, una forma sencilla de obtener la figura es utilizando estos comandos:
unset specular
unset depth_cue
set ambient, 0.6
set ray_trace_mode, 1
set ray_trace_depth_factor, 1
set ray_trace_disco_factor, 1
Luego de renderizar mediante el comando ray, debería aparecer algo similar a esto:
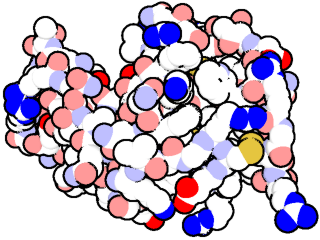
Es posible que sea necesario ajustar el parámetro ambient para evitar la aparición del sombreado en las esferas.3 Además, dependiendo del tamaño de la renderización quizás haya que modificar el valor de ray_trace_gain para alterar el grosor de los bordes.
Una manera más complicada
Bonnie Scott publicó hace un tiempo un tutorial en el cual ocupa dos metodologías para obtener figuras al estilo Goodsell. Una usa Chimera, mientras que la otra emplea una combinación de ePMV y Cinema 4D. Ambas aproximaciones consisten en renderizar por separado colores, sombras y bordes, para luego unirlas usando algún editor gráfico como Photoshop. Voy a adaptar esa estrategia para PyMOL. Este método es más engorroso que el anterior, pero permite un mayor control sobre el resultado final.
En este caso voy a crear una imagen de la estructura de una proteína anticongelante del escarabajo Rhagium inquisitor (PDB ID: 4DT5). Primero hay que cargar la estructura y preparar la escena:
hide all
remove solvent
remove hetatm
show spheres, polymer
unset depth_cue
unset specular
unset ray_shadow
set ambient, 1
bg_color black
Luego se renderizan:
-
La máscara alfa de la proteína completa.
color white, polymer ray 1000, 1000 save protein.png -
La máscara alfa de los átomos de nitrógeno y oxígeno.
color black, polymer color white, name N*+O* ray 1000, 1000 save n+o.png -
Las sombras.
bg_color white util.ray_shadows('black') set reflect, 1 set reflect_power, 0 color white, polymer ray 1000, 1000 save shadows.png -
Y, finalmente, el z-buffer4.
bg_color black set depth_cue unset ray_shadow set fog_start, 0 clip atoms, 2, all color white, polymer ray 1000, 1000 save z-buffer.png
Con lo que se obtienen las siguientes imágenes:

Estas deben ser abiertas como capas en Photoshop, GIMP, etc. y procesadas de la siguiente manera:
- La máscara alfa de la proteína completa y la de los nitrógenos y oxígenos se transforman en una máscara rápida y se rellenan del color deseado.
- Este paso es opcional: Sobre la imagen de las sombras se aplica desenfoque gaussiano y la máscara alfa de la proteína completa para que las sombras no sobrepasen el borde de la proteína.
- Del z-buffer se pueden derivar los bordes usando, por ejemplo, el operador Sobel en GIMP (Filtros ‣ Detectar bordes ‣ Arista) o el filtro “Hallar bordes” de Photoshop (Filtro ‣ Estilizar ‣ Hallar bordes). Luego se deben ajustar los niveles o curvas para eliminar los bordes falsos del interior de las esferas. Se pueden encontrar más detalles en este tutorial.
Al finalizar estos pasos, se obtienen las siguientes capas:

La cuales se juntan (los bordes y sombras van apilados en modo Multiplicar) para obtener la figura final:
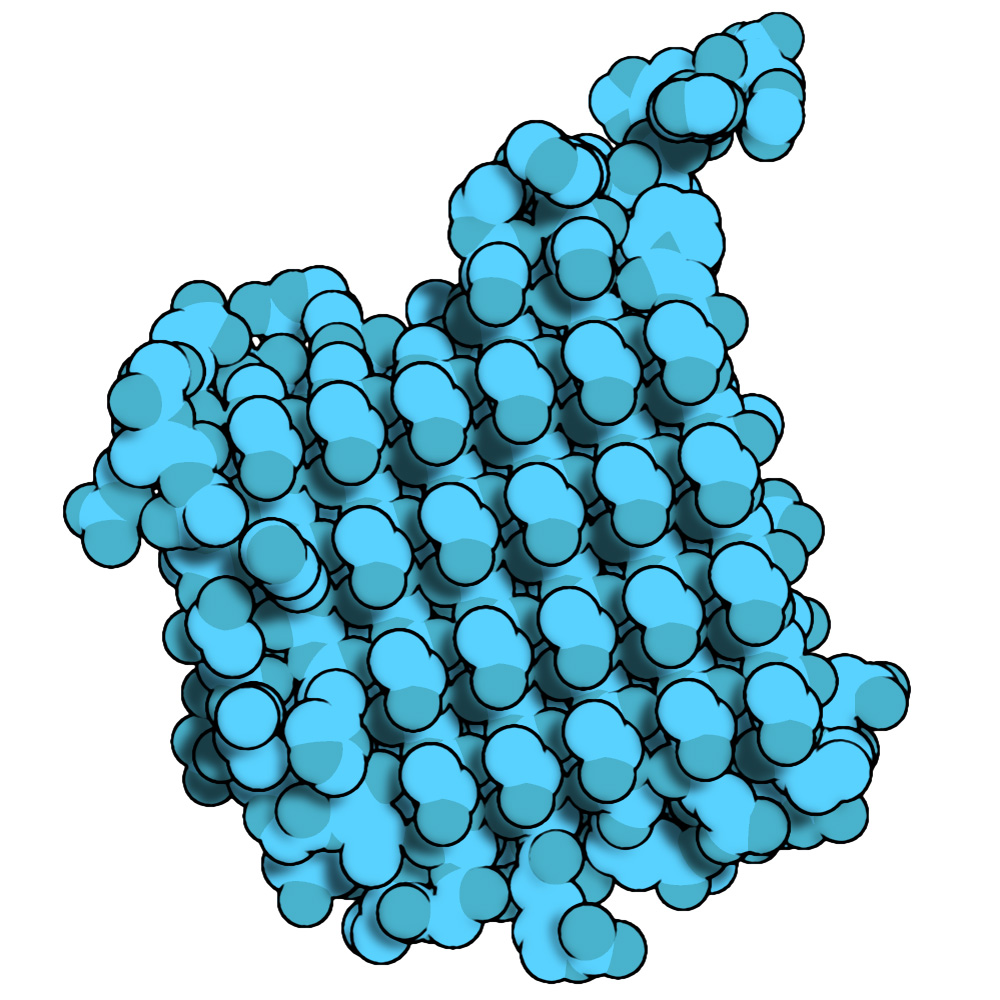
Como pueden ver, esta estructura contiene un arreglo muy ordenado de treoninas, encargadas de coordinar moléculas de agua, logrando detener el crecimiento de los cristales de hielo que aparecen en el ambiente celular.
Más información
Si quieren ver más sobre la obra de David Goodsell, revisen su página web, la sección Molecule of the Month en el PDB, o su libro The Machinery of Life, el cual contiene varias de sus ilustraciones.
-
Por ejemplo: Usando shaders GLSL con PyMOL y VMD. Un tutorial en Chemistry Stack Exchange con VMD, Blender y Photoshop. Tutoriales en PyMOL, Chimera y PMV. También existen programas como Qutemol y Zink!. ↩
-
Esta combinación aparece por última vez en MoTM en octubre del 2005. Una paleta similar pero, en mi opinión, más moderna, apareció en la entrada de junio del 2013. Actualmente Goodsell utiliza en la mayoría de las ocasiones colores arbitrarios para el carbono y un tono más oscuro para nitrógeno, oxígeno y azufre. ↩
-
El parámetro
ambientcontrola la cantidad de luz ambiente en la escena. Acepta valores entre 0 y 1, y por defecto es igual a 0.1. ↩ -
A grandes rasgos, el z-buffer es una representación de la profundidad en la que se encuentran ubicados los objetos de una escena. ↩